Copy Number Variations
Genamplificaties kunnen met meerdere technieken aangetoond worden waarbij het van de vraagstelling afhangt welke techniek het best gebruikt kan worden. Indien de amplificatie status van meerdere genen gewenst is, kan Next Generation Sequencing of de SNP array gebruikt worden. Als er slechts een enkel gen van belang is, kan een meer gerichte analyse uitgevoerd worden zoals FISH (bijvoorbeeld HER2/neu voor borsttumoren) of MLPA (bijvoorbeeld MDM2 voor liposarcomen).

Voorbeeld van een FISH voor gen amplificatie, waarbij met het groene signaal HER2/neu amplificatie is weergegeven
SNP array (tumorklonaliteitsanalyse)
Voor tumorklonaliteit vraagstukken kan indien er op basis van tumortype weinig mutaties verwacht worden met Next Generation Sequencing een SNP array analyse uitgevoerd worden. Deze analyse is gebaseerd op het feit dat verschillende tumoren verschillende chromosomale afwijkingen zullen hebben. Met name breukpunten van chromosoomarmen zijn dusdanig specifiek dat deze gebruikt kunnen worden om te beoordelen of 2 tumoren dezelfde klonale oorsprong hebben. Hiervoor wordt de Illumina 850k beadchip gebruikt waarmee 850.000 Single Nucleotide Polymorphisms (SNPs) geanalyseerd worden. Heterozygote SNPs verschillen tussen het maternale en paternale allel en de verhouding tussen beide allelen is in normaal weefsel 50:50. Indien er sprake is van een gain of een loss van een van beide chromosomen, zal de verhouding tussen beide allelen gaan verschuiven. Op basis van het patroon van de chromosomale afwijkingen en de locatie van de chromosomale breukpunten kan uitspraak gedaan worden over een eventueel klonaal verwantschap van verschillende tumoren.
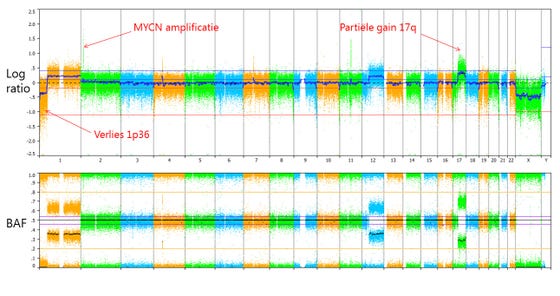
Voorbeeld van een SNP-array analyse van een neuroblastoom met verlies van chromosoom 1p36, amplificatie van MYCN en gedeeltelijke verhoging van chromosoom 17q.
