Methylatie analyses
Methylatie array
Uit recent onderzoek is gebleken dat verschillende types hersentumoren een ander methylatie profiel hebben. Methylatie is de toevoeging van een CH3 groep aan één van de nucleotiden in het DNA waardoor genexpressie geremd wordt. Doordat dit methylatie profiel zo specifiek is kan het fungeren als een tumor specifieke vingerafdruk waarmee de tumor geïdentificeerd kan worden, met name als dat op basis van histologie lastig is. Hiervoor wordt de Illumina EPIC 850k beadchip gebruikt waarmee 850.000 verschillende methylatie sites in één experiment geanalyseerd worde, gecombineerd met een analyse methode die is ontwikkeld door de Universiteit van Heidelberg. Daarnaast wordt met behulp van deze techniek informatie verkregen over copy-number variations (CNVs) en de methylatie status van genen van interesse zoals MGMT.
Momenteel kan deze assay ook uitgevoerd voor de classificatie van sarcomen.
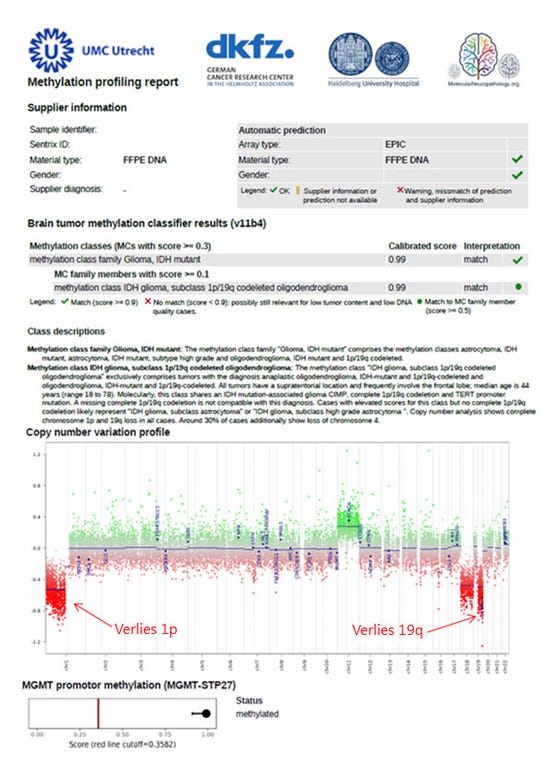
Voorbeeld van een methylatie array rapport van een oligodendroglioom met verlies van chromosoom 1p en 19q en methylatie van MGMT.
Promotor hypermethylatie
De methylatie status van een enkel gen kan geanalyseerd worden met behulp van Methylatie Specifieke Multiplex Ligation-dependent Probe Amplification (MS-MLPA). Met behulp van methylatie specifieke restrictie enzymen wordt onderscheid gemaakt tussen gemethyleerd en ongemethyleerd DNA. De MS-MLPA wordt uitgevoerd voor de analyse van MGMT methylatie voor glioblastomen (tevens wordt de IDH mutatie status bepaald) en van MLH1 voor Lynch Syndroom vraagstellingen.
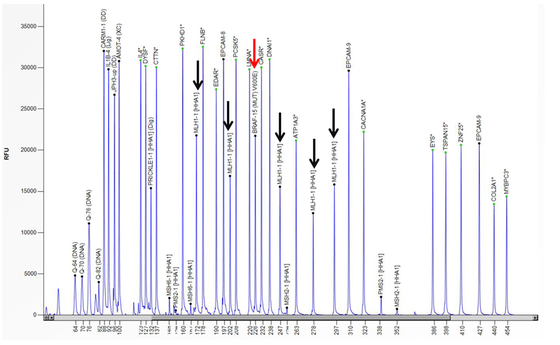
Voorbeeld van een electropherogram van MS-MLPA voor MLH1 van een patiënt met MLH-1 methylatie en een BRAF V600E mutatie. De rode pijl toont de piek van de probe voor de BRAF V600E mutatie, de zwarte pijlen tonen de pieken van de MLH-1 probes.
